Los científicos han descubierto que una bacteria puede usar fragmentos de antiguos virus como escudo contra nuevas infecciones. El hallazgo, liderado por un equipo internacional y publicado en Nucleic Acids Research, muestra cómo un “virus fósil” integrado en el ADN de Escherichia coli puede despertar para protegerla de ataques virales modernos. Estos restos, llamados cripto‐profagos, son virus tan antiguos que perdieron su capacidad de reproducirse, pero conservaron genes útiles que la bacteria ha aprendido a aprovechar a su favor.
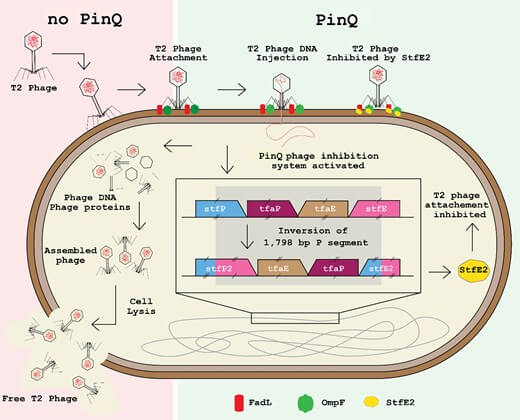
En este caso, los investigadores analizaron una enzima llamada PinQ, una recombinasa, es decir, una proteína capaz de cortar y reordenar fragmentos de ADN. Aunque estos genes solían considerarse piezas inactivas o sin función, el estudio demostró que PinQ tiene un papel crucial en la defensa de la bacteria frente al fago T2, un virus que infecta células bacterianas. Cuando el T2 se acerca, la bacteria activa a PinQ, que reorganiza una pequeña sección de su propio ADN para generar nuevas proteínas de defensa.
Este proceso es tan antiguo como sofisticado. En lugar de destruir al invasor, la bacteria simplemente impide que el virus pueda adherirse a su superficie, cortando de raíz el ciclo de infección. Es la primera vez que se observa un sistema antiviral activado por recombinación genética, un tipo de “defensa molecular” que transforma el ADN para bloquear la entrada del virus.

Cómo funciona el sistema que bloquea al virus
En condiciones normales, PinQ permanece inactivo, como un interruptor apagado. Pero cuando el virus intenta infectar, la enzima invierte un fragmento de ADN dentro de la bacteria, lo que provoca la creación de dos proteínas completamente nuevas. Una de ellas, llamada StfE2, se adhiere a la membrana de la célula bacteriana y actúa como una barrera física que impide al virus encontrar sus receptores habituales, las proteínas FadL y OmpF, que el fago T2 usa para engancharse.
Esa inversión del ADN equivale a “dar la vuelta” a una pieza genética, pero en lugar de dañar a la célula, la transforma en una herramienta de defensa. Los investigadores comprobaron que, tras esta reconfiguración, el fago T2 ya no puede adherirse ni reproducirse dentro de la bacteria, lo que la hace completamente resistente a la infección. Además, al eliminar el gen PinQ, la bacteria vuelve a ser vulnerable, confirmando el papel protector del sistema.
Este hallazgo revela algo que hasta ahora se había pasado por alto: las recombinasas no son simples vestigios de antiguos virus, sino defensas activas que pueden encenderse cuando la bacteria las necesita.
Durante años, los científicos las consideraban marcadores genéticos sin relevancia, pero este estudio demuestra que son piezas fundamentales del arsenal antiviral bacteriano.
Una defensa con consecuencias evolutivas
El equipo también observó que, una vez desaparece el virus, la bacteria invierte nuevamente el proceso. El segmento de ADN vuelve a su posición original, lo que indica que esta defensa no es permanente, sino reversible, un detalle importante porque mantenerla activada tiene un coste biológico.
Las bacterias con el ADN invertido crecen más lentamente en ciertos medios, lo que sugiere que este sistema solo se activa cuando hay peligro real.
El estudio confirma que la activación de PinQ no solo depende del ataque viral, sino que también forma parte de un equilibrio evolutivo: las bacterias prefieren “apagar” la defensa cuando ya no es necesaria para ahorrar energía. Esta flexibilidad podría explicar por qué mecanismos similares se conservan en tantas especies bacterianas, incluso en aquellas que han perdido la capacidad de producir virus.
Los investigadores encontraron versiones del gen PinQ en docenas de bacterias y virus, incluidos patógenos que afectan a humanos y animales. Esto sugiere que el sistema de defensa basado en recombinasas es mucho más común de lo que se pensaba y que podría representar una estrategia evolutiva extendida para enfrentar las infecciones virales en el mundo microbiano.
La respuesta del virus: una carrera por sobrevivir
El fago T2, por supuesto, no se queda quieto. Al enfrentar repetidamente el sistema de defensa de PinQ, algunos virus logran adaptarse. Tras varios ciclos de exposición, los científicos detectaron que el T2 desarrolla mutaciones en su proteína de adhesión, Gp38, la responsable de identificar y unirse a la célula. Con un pequeño cambio en un aminoácido, el virus logra escapar de la barrera creada por StfE2 y retoma su capacidad de infección.
Este comportamiento confirma que las bacterias y los virus mantienen una guerra constante a nivel molecular. Mientras una modifica su ADN para bloquear la entrada, el otro cambia su estructura para abrir una nueva puerta.
Según los autores, entender esta dinámica puede ayudar a diseñar tratamientos más efectivos basados en fagos —virus que atacan bacterias—, una alternativa que cobra relevancia ante la creciente resistencia a los antibióticos.
Además, la investigación demuestra que estos enfrentamientos no solo son un fenómeno de laboratorio. Los genomas de muchas bacterias actuales conservan huellas de antiguas batallas con virus, lo que refuerza la idea de que los profagos, aunque inactivos, siguen participando activamente en la evolución bacteriana.
Implicaciones para la salud y la biotecnología
El descubrimiento abre una nueva ventana sobre cómo funcionan las defensas naturales de las bacterias. Comprender estos mecanismos puede ser clave para desarrollar tratamientos más precisos contra infecciones resistentes a antibióticos, ya que los fagos podrían aprovecharse sin interferir con los sistemas de defensa del huésped. Si se logra controlar cuándo y cómo se activa una recombinasa como PinQ, sería posible fortalecer bacterias beneficiosas o diseñar terapias más seguras basadas en virus.
También existen implicaciones en la industria alimentaria. Las bacterias que se usan para fermentar alimentos —como las del yogur o el queso— son vulnerables a infecciones por fagos que arruinan los cultivos. Conocer estos mecanismos naturales de defensa podría mejorar la seguridad y estabilidad de los procesos de fermentación, reduciendo pérdidas y evitando el uso de aditivos químicos.
Referencias
- Kirigo, J., Huelgas‐Méndez, D., Tomás, M., Benedik, M. J., García‐Contreras, R., & Wood, T. K. (2025). Adsorption of phage T2 is inhibited due to inversion of cryptic prophage DNA by the serine recombinase PinQ. Nucleic Acids Research, 53(19), gkaf1041. doi: 10.1093/nar/gkaf1041




